
Fusions du gène FGFR2 (récepteur 2 du facteur de croissance des fibroblastes) dans le CCA intra-hépatique
Récepteurs des facteurs de croissance des fibroblastes (FGFRs) et altérations génomiques
- Les récepteurs FGFR font partie d’une famille de récepteurs à activité tyrosine kinase.1,2 La voie de signalisation FGFR joue un rôle central dans de multiples processus cellulaires, tels que la prolifération, la migration et la survie cellulaire1,2
- Les altérations génomiques des gènes FGFR sont des drivers oncogéniques dans un certain nombre de cancers, incluant notamment le CCA intra-hépatique, le carcinome urothélial et les néoplasies myéloïdes/lymphoïdes1,3,4
- Des amplifications, des mutations et des gènes de fusion ont été observés dans tous les
sous-types de récepteurs FGFR (FGFR1–4).5
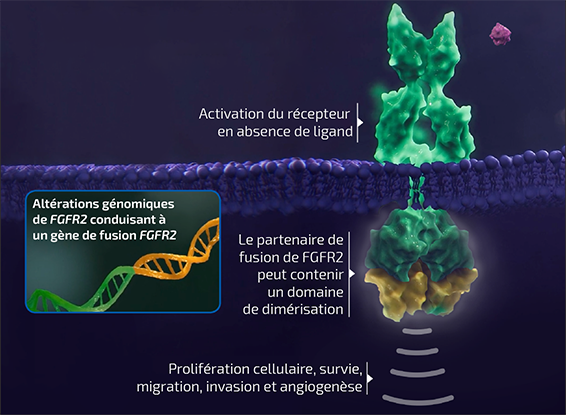
Les réarrangements chromosomiques impliquant FGFR2 – conduisant à la création d’une
protéine de fusion oncogénique – ont été fréquemment identifiés dans les CCA intra-hépatique6
- Les gènes de fusions sont un type d’altération génomique ou deux gènes indépendants ou deux fragments de gènes se retrouvent associés et conduisent à la formation d’un gène chimère8
- Les protéines de fusion potentiellement oncogéniques sont souvent issues de fusion génétique impliquant une gamme de gènes partenaires différents7
FGFR et altérations génomiques
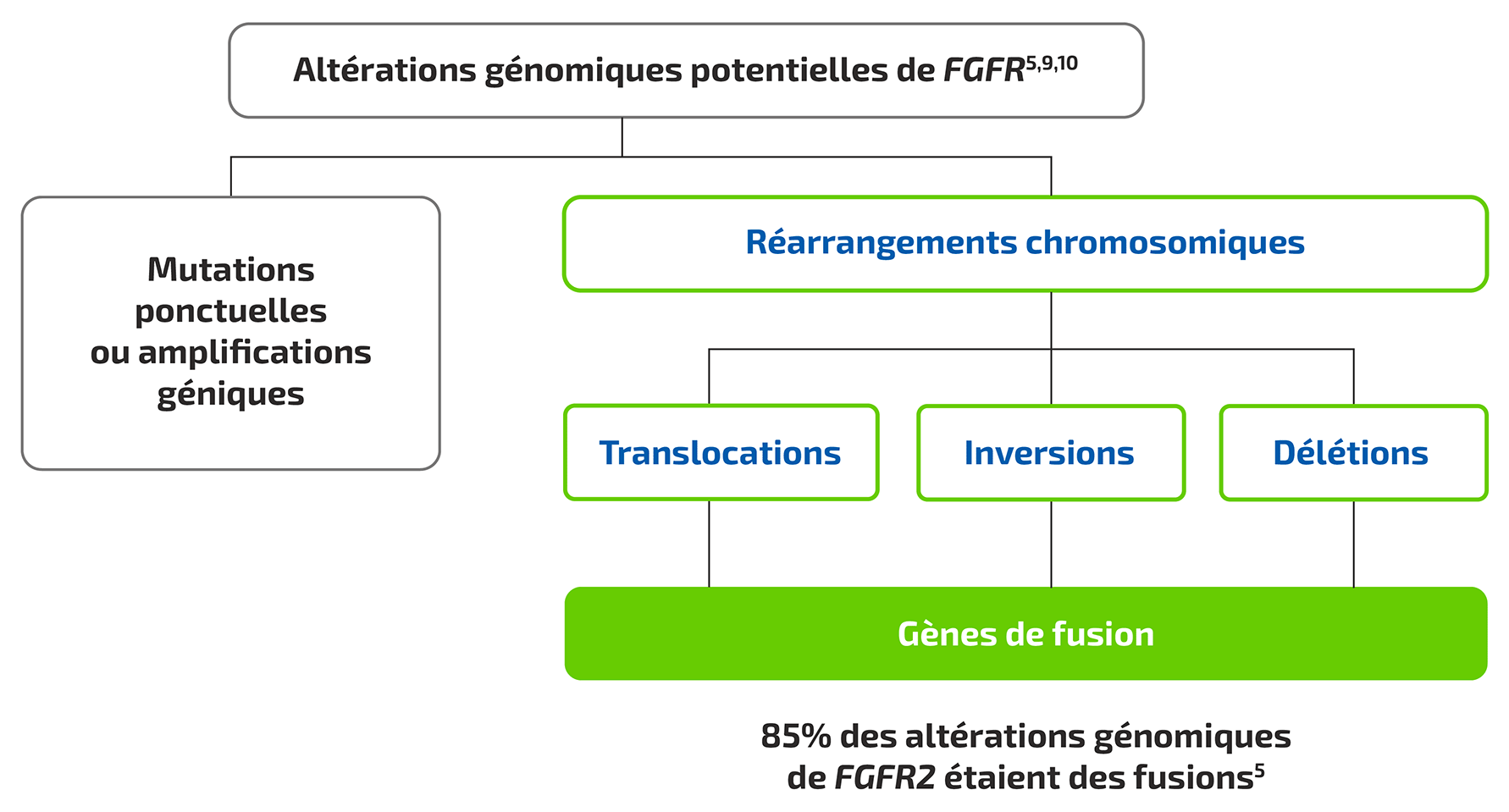
Figure adaptée de Jain A, et al. 2018,5 Lowery MA, et al. 2018,9 et Shibata T, et al. 2018.10

 Avantages
Avantages
 Inconvénients
Inconvénients
