Altérations génomiques dans le CCA intra-hépatique
Des altérations génomiques potentiellement "actionnables" ont été identifiées chez environ 40 % des patients atteints de CCA intra-hépatique, permettant d’établir un tableau plus personnalisé de la biologie de la maladie de chaque patient1
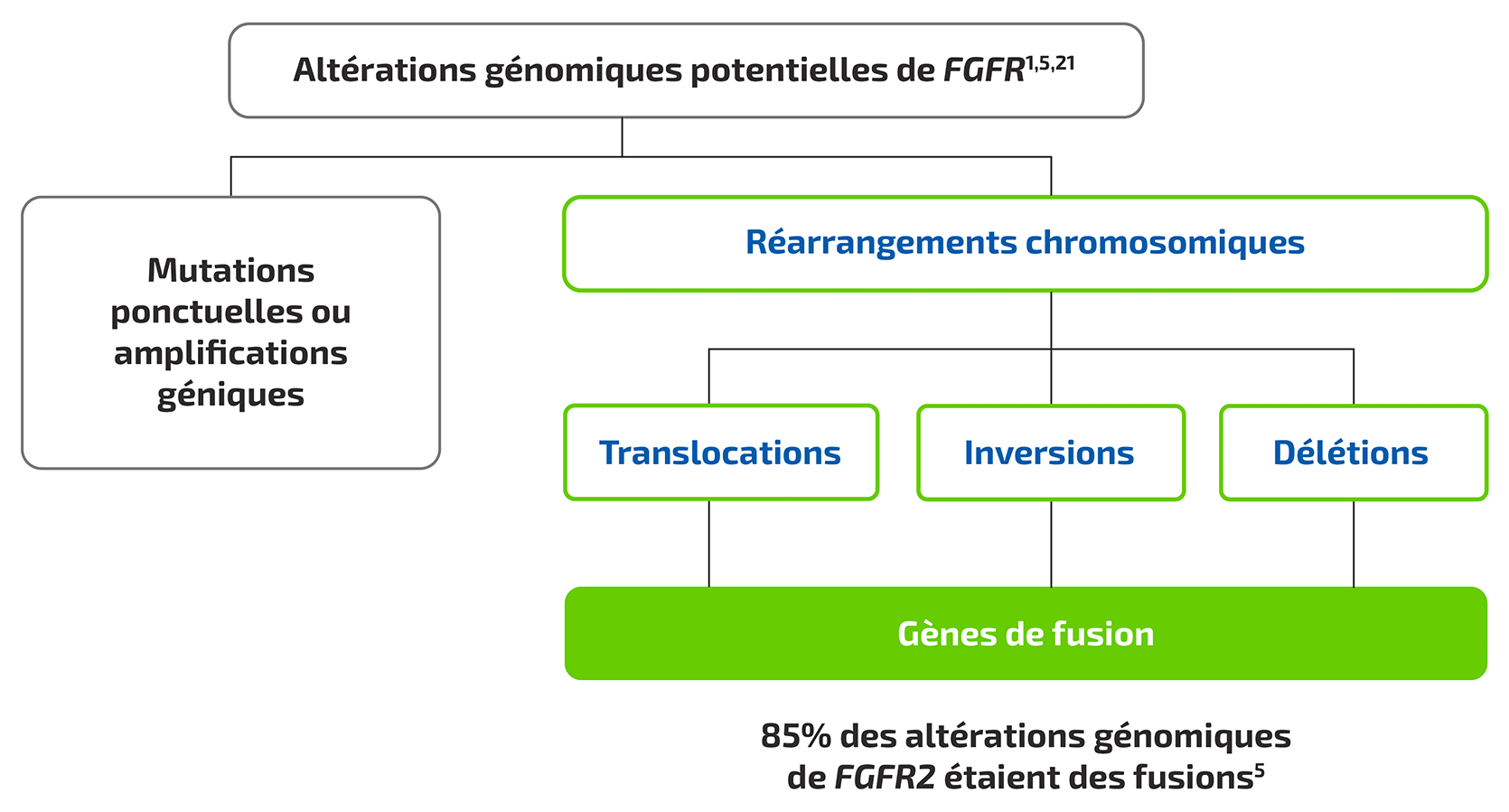
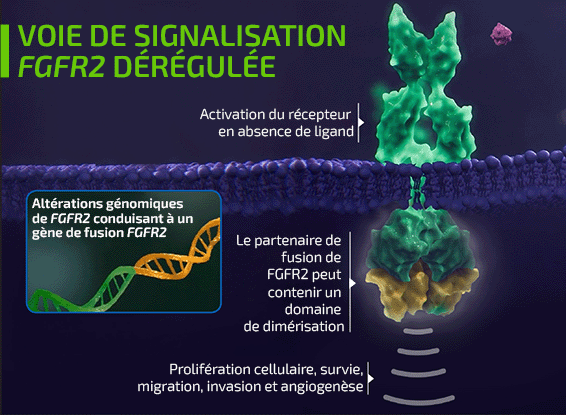
- Deux des altérations génomiques les plus fréquentes associées au CCA intra-hépatique sont les mutations de l’isocitrate déshydrogénase (IDH) et les fusions du récepteur 2 du facteur de croissance des fibroblastes (FGFR2)1
Les altérations génomiques potentiellement actionnables
sont nombreuses dans le CCA
intra-hépatique1*

*Basé sur une analyse rétrospective de 6 130 patients diagnostiqués avec un CCAi à partir de la base de données FoundationCORE qui ont reçu un séquençage de panel diagnostique sur la plateforme FoundationOne. Les chercheurs ont analysé des mutations ponctuelles, des fusions/réarrangements et des variations du nombre de copies dans plus de 300 gènes liés aux tumeurs, et la plupart des participants de l'étude avaient des données disponibles sur le statut TMB et MSI.1
amp, amplification ; BRAF, proto-oncogène B-Raf ; BRCA1/2, gène du cancer du sein 1/2 ; ERBB2, récepteur erb-b2 tyrosine kinase 2 ; CCA, cholangiocarcinome ; FGFR2, récepteur 2 du facteur de croissance des fibroblastes ; IDH1/2, isocitrate déshydrogénase 1/2; KRAS, homologue de l’oncogène viral du sarcome de rat Kirsten ; MDM2, souris double minute 2 homologue 2 ; MET, proto-oncogène de transition épithéliale mésenchymateuse ; MSI, instabilité des microsatellites ; MSI-H, instabilité des microsatellites élevée ; mut, mutation ; TMB, charge mutationnelle tumorale.
Figure adaptée de Kendre G. 2023.1