
Fusiones del receptor 2 del factor de crecimiento de fibroblastos (FGFR2) en el colangiocarcinoma intrahepático (CCAi)
Alteraciones genómicas en los receptores del factor de crecimiento de fibroblastos (FGFR)
- Los FGFR son una familia receptores con actividad tirosina cinasa1,2
Las vías de señalización del FGFR desempeñan un papel central en múltiples procesos celulares, incluida la proliferación, migración y supervivencia celulares1,2
- Las alteraciones en los genes FGFR han surgido como impulsores de la génesis tumoral en varios tipos de cáncer, como el CCAi, el carcinoma urotelial, las neoplasias mieloides/linfoides y otras neoplasias malignas1,3,4
- Se han observado amplificaciones, mutaciones y fusiones del FGFR en todos los
subtipos del FGFR (FGFR1–4).5
En el CCAi se han identificado con frecuencia reordenaciones cromosómicas que implican al
FGFR2, lo que resulta en la creación de proteínas de fusión oncogénicas6
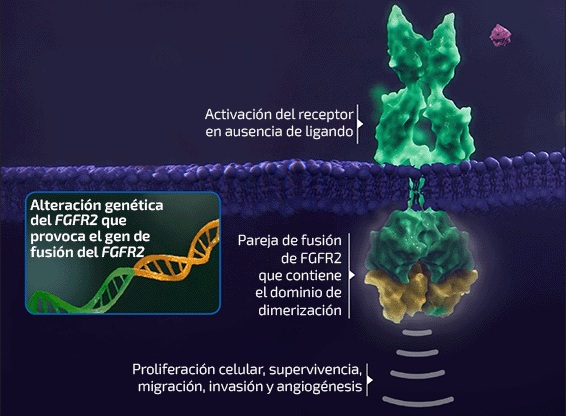
- Las fusiones génicas son un tipo de alteración genómica en la que dos genes independientes o porciones de genes se yuxtaponen, lo que da lugar a un gen híbrido7,8
- El desarrollo de proteínas de fusión con potencial oncogénico puede deberse a acontecimientos de fusión génica que implican una serie de parejas de fusión diferentes
Alteraciones genómicas del FGFR
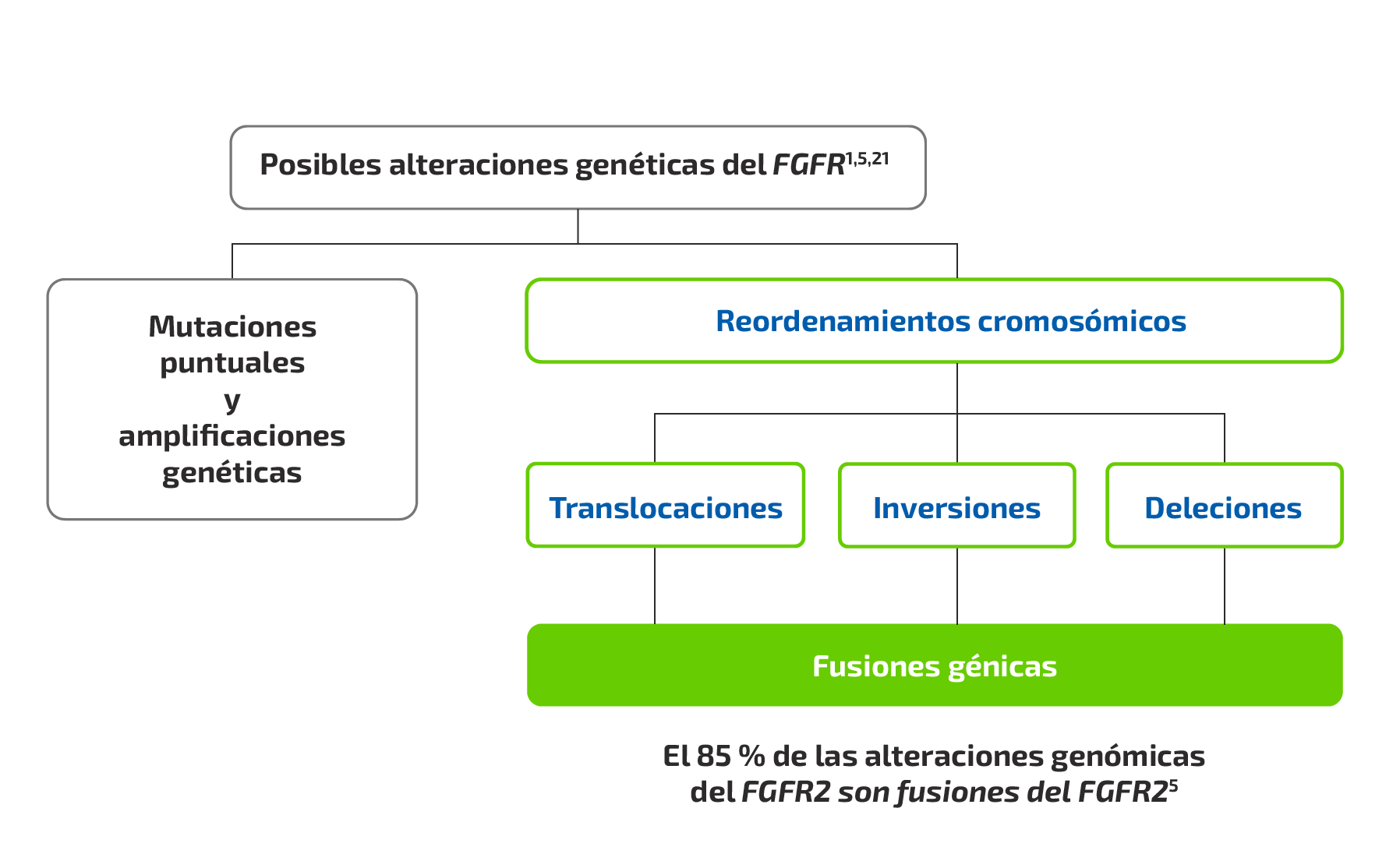
Figura basada en Jain A, et al. 2018,5 Lowery MA, et al. 2018,9 y Shibata T, et al. 2018.10


 Ventajas
Ventajas
 Retos
Retos
