
Fusionen des Fibroblasten-Wachstumsfaktor-Rezeptors 2 (FGFR2) beim intrahepatischen Cholangiokarzinom (iCCA)
Genveränderungen in Fibroblasten-Wachstumsfaktor-Rezeptoren (FGFR)
- FGFR sind eine Familie von Rezeptortyrosinkinasen.1,2
Die FGFR-Signalwege spielen eine zentrale Rolle bei zahlreichen zellulären Prozessen wie Proliferation, Migration dem Überleben von Zellen1,2
- Veränderungen in FGFR-Genen haben sich als tumorgene Treiber bei Krebsarten wie iCCA, Urothelkarzinomen, myeloiden/lymphoiden Neoplasmen und anderen malignen Erkrankungen erwiesen1,3,4
- FGFR-Amplifikationen, -Mutationen und -Fusionen werden bei allen FGFR-Subtypen
(FGFR1–4) beobachtet.5
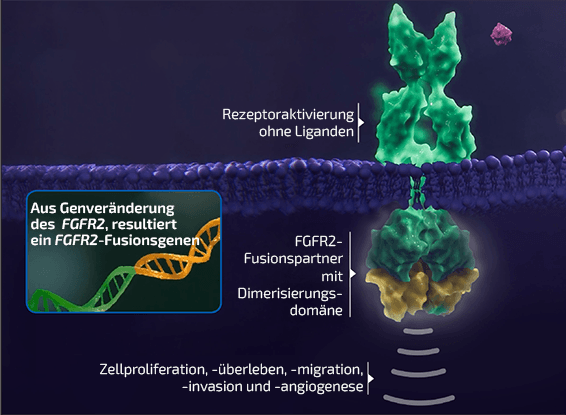
Chromosomale Rearrangements unter Beteiligung von FGFR2 – die zur Bildung onkogener
Fusionsproteine führen – sind bei iCCA häufig zu finden6
- Bei Genfusionen handelt es sich um Genveränderungen, bei denen sich zwei unabhängige Gene oder Teile von Genen miteinander verbinden, was zu einem hybriden Gen führt7,8
- Aus Genfusionsereignissen unter Beteiligung einer Reihe verschiedener Partnergene können sich Fusionsproteine mit onkogenem Potenzial entwickeln7
Genomische FGFR-Veränderungen
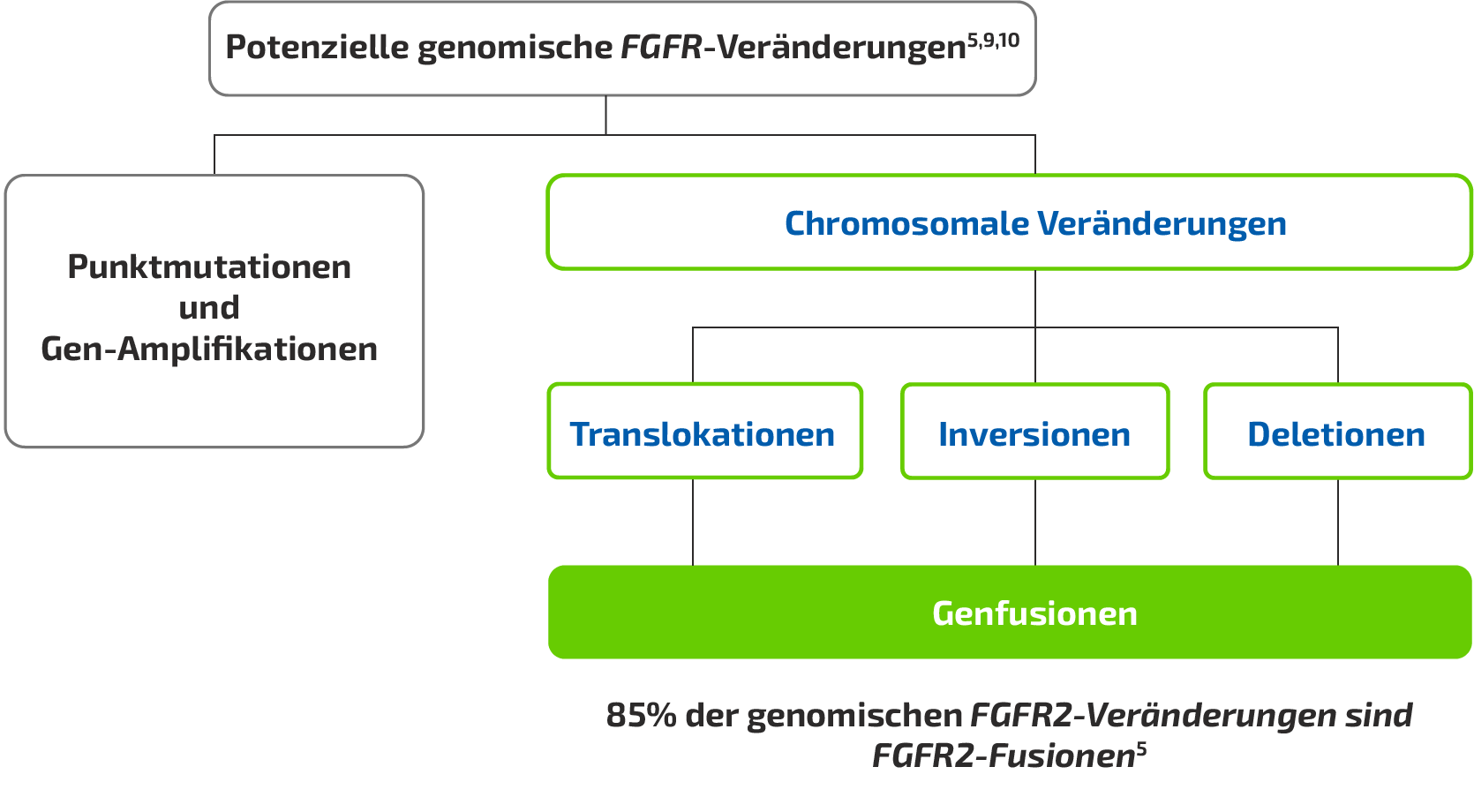
Abbildung basiert auf Jain A, et al. 2018,5 Lowery MA, et al. 2018,9 und Shibata T, et al. 201810

 Vorteile
Vorteile
 Nachteile
Nachteile
